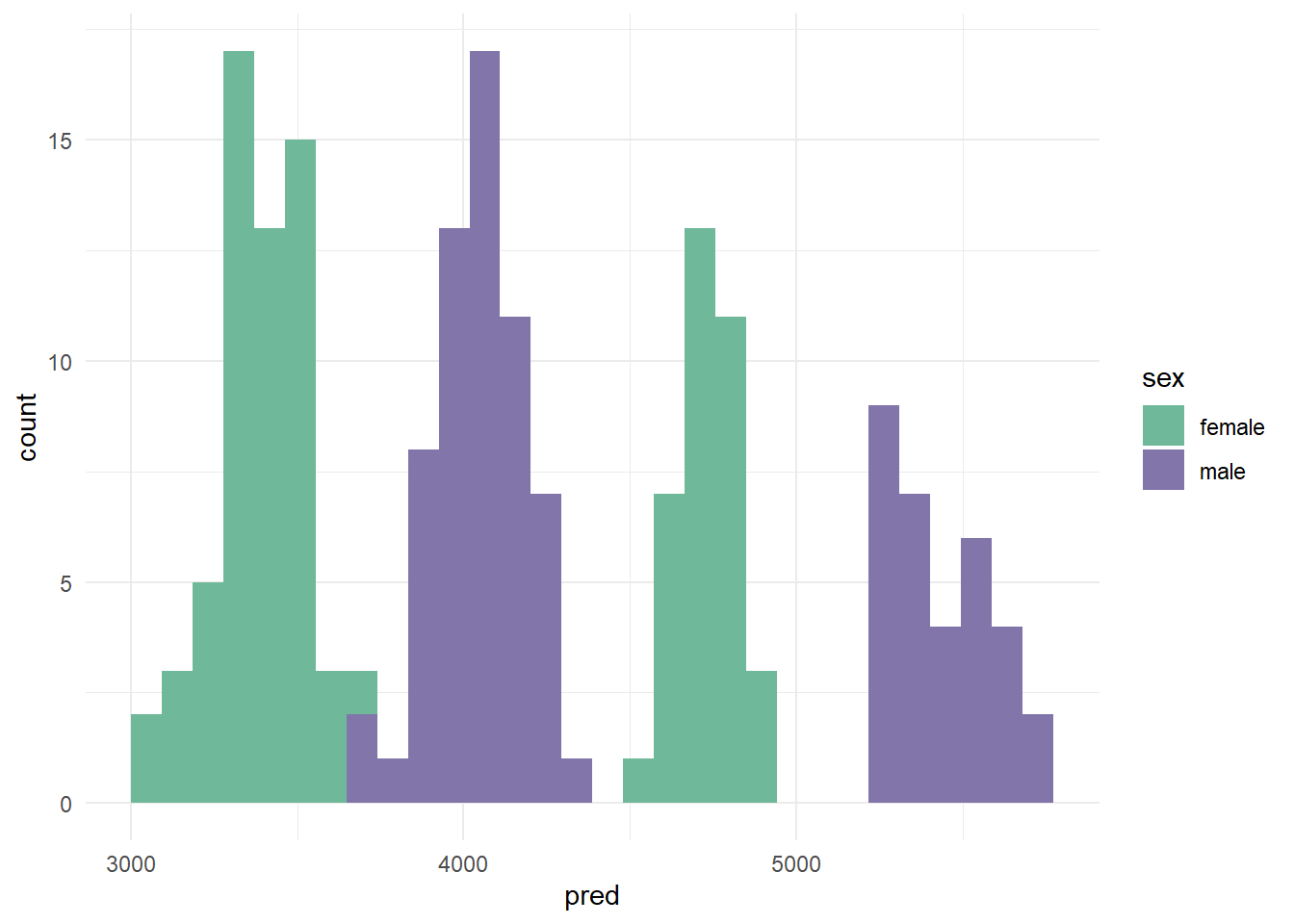Einleitung
Im Folgenden wird die Vorhersagemodellierung noch einmal genauer behandelt. Um Overfitting zu vermeiden wird der Train-Datensatz aufgeteilt.
Pakte laden
library (rbthemes)library (tidymodels) #für Auteilung der Daten reicht es, das Paket rsample zu laden
── Attaching packages ────────────────────────────────────── tidymodels 1.2.0 ──
✔ broom 1.0.5 ✔ recipes 1.0.10
✔ dials 1.2.1 ✔ rsample 1.2.1
✔ dplyr 1.1.4 ✔ tibble 3.2.1
✔ ggplot2 3.5.1 ✔ tidyr 1.3.1
✔ infer 1.0.7 ✔ tune 1.2.1
✔ modeldata 1.3.0 ✔ workflows 1.1.4
✔ parsnip 1.2.1 ✔ workflowsets 1.1.0
✔ purrr 1.0.2 ✔ yardstick 1.3.1
── Conflicts ───────────────────────────────────────── tidymodels_conflicts() ──
✖ purrr::discard() masks scales::discard()
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()
✖ recipes::step() masks stats::step()
• Learn how to get started at https://www.tidymodels.org/start/
── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
✔ forcats 1.0.0 ✔ readr 2.1.5
✔ lubridate 1.9.3 ✔ stringr 1.5.1
── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
✖ readr::col_factor() masks scales::col_factor()
✖ purrr::discard() masks scales::discard()
✖ dplyr::filter() masks stats::filter()
✖ stringr::fixed() masks recipes::fixed()
✖ dplyr::lag() masks stats::lag()
✖ readr::spec() masks yardstick::spec()
ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errors
Attache Paket: 'palmerpenguins'
Das folgende Objekt ist maskiert 'package:modeldata':
penguins
<- penguins
Daten importieren und aufteilen
Die folgende Aufteilung wird im Vorhinein vom Dozenten vorgenommen. In der Prüfung bekommt ihr d_train und d_test als Resultat des folgenden Codes:
<- penguins_data %>% drop_na () %>% mutate (id = row_number ()) %>% select (id, everything ())set.seed (123 )<- initial_split (penguins_data, prop = 0.7 )<- training (train_test_split)<- testing (train_test_split)
Hier wird die Aufteilung des Train-Samples vorgenommen. Auf d_train1 werden Modelle trainiert, um dann auf d_train_test angewandt zu werden. So kann man sicherstellen, dass das Modell nicht nur die Daten, die es kennt, gut vorhersagt, sondern auch unbekannte Daten. Es gilt zu beachten, dass am Ende ein Modell abgegeben werden sollte, das auf dem kompletten ursprünglichen Train-Sample trainiert wurde.
<- initial_split (d_train, prop = 0.8 )<- training (dtrain_split)<- testing (dtrain_split)
EDA
%>% group_by (sex) %>% summarise (mean (body_mass_g))
female 3875.210 male 4564.912
Vorhersagemodellierung
<- lm (body_mass_g ~ I (bill_depth_mm^ 2 ) + species + flipper_length_mm+ bill_length_mm + sex, data = d_train1)summary (lm1)
Call:
lm(formula = body_mass_g ~ I(bill_depth_mm^2) + species + flipper_length_mm +
bill_length_mm + sex, data = d_train1)
Residuals:
Min 1Q Median 3Q Max
-783.22 -171.88 -31.76 180.76 726.45
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -617.0111 710.7614 -0.868 0.386503
I(bill_depth_mm^2) 1.8207 0.7431 2.450 0.015245 *
speciesChinstrap -351.9506 114.5383 -3.073 0.002452 **
speciesGentoo 968.2548 167.9924 5.764 3.54e-08 ***
flipper_length_mm 13.2436 3.8255 3.462 0.000671 ***
bill_length_mm 26.2270 10.4342 2.514 0.012834 *
sexmale 395.7132 66.2268 5.975 1.21e-08 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 292.3 on 179 degrees of freedom
Multiple R-squared: 0.8696, Adjusted R-squared: 0.8652
F-statistic: 198.9 on 6 and 179 DF, p-value: < 2.2e-16
<- lm (body_mass_g ~ flipper_length_mm + bill_depth_mm + bill_length_mm + sex, data = d_train1)summary (lm2)
Call:
lm(formula = body_mass_g ~ flipper_length_mm + bill_depth_mm +
bill_length_mm + sex, data = d_train1)
Residuals:
Min 1Q Median 3Q Max
-921.34 -262.15 0.28 253.06 926.54
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -1543.865 883.423 -1.748 0.0822 .
flipper_length_mm 36.896 3.440 10.726 < 2e-16 ***
bill_depth_mm -95.565 22.281 -4.289 2.91e-05 ***
bill_length_mm -7.090 7.042 -1.007 0.3154
sexmale 585.857 74.296 7.885 2.85e-13 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 358.6 on 181 degrees of freedom
Multiple R-squared: 0.8015, Adjusted R-squared: 0.7972
F-statistic: 182.8 on 4 and 181 DF, p-value: < 2.2e-16
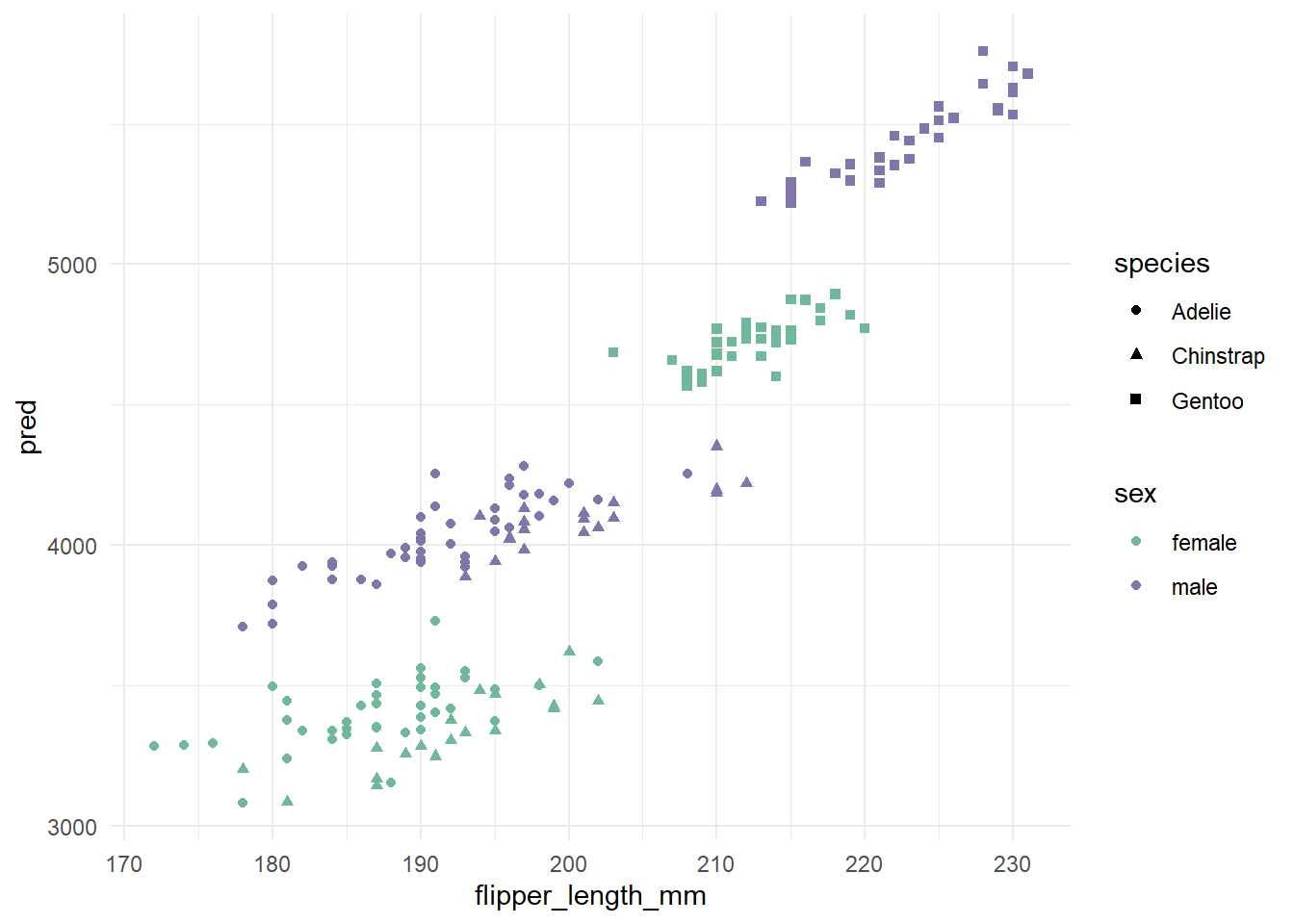
<- d_train1 %>% mutate (pred = predict (lm1, newdata = d_train1))
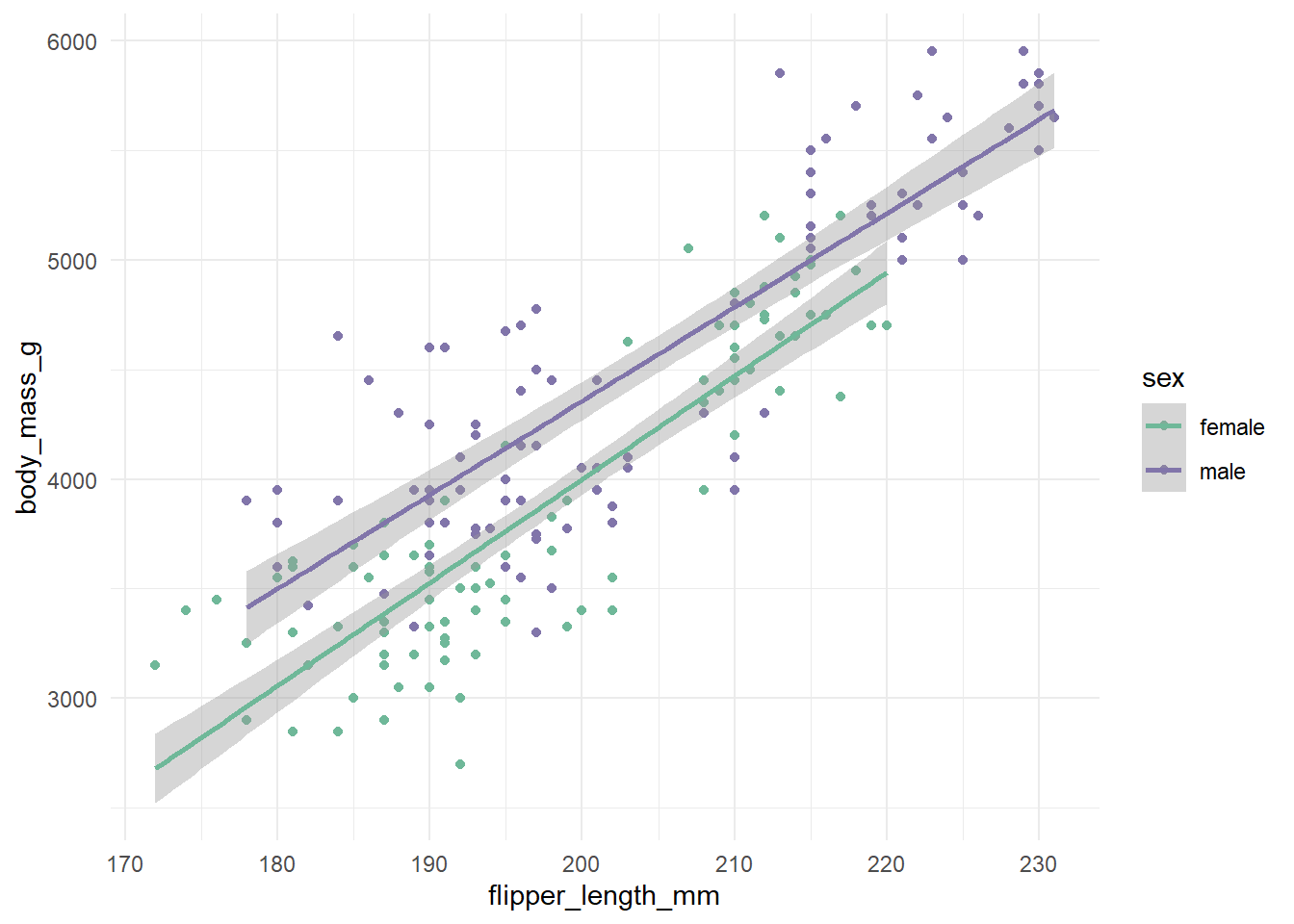
ggplot (data = d_train1, aes (x = flipper_length_mm, y = body_mass_g, color = sex)) + geom_point () + geom_smooth (method = "lm" ) +
`geom_smooth()` using formula = 'y ~ x'
ggplot (data = d_train1, aes (x = flipper_length_mm, y = pred)) + geom_point (aes (color = sex, shape = species)) +
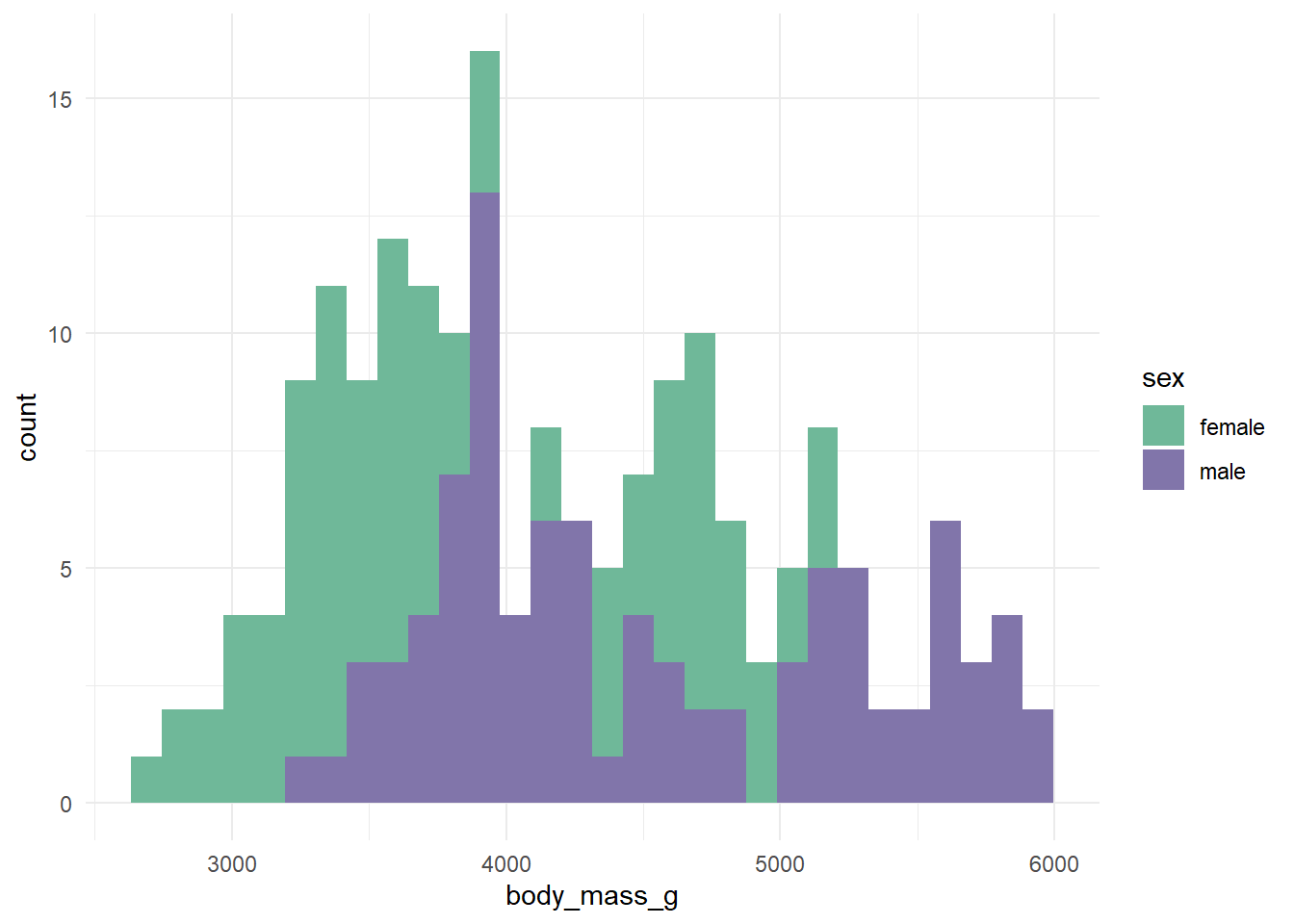
ggplot (data = d_train1, aes (x = body_mass_g, fill = sex)) + geom_histogram () +
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
ggplot (data = d_train1, aes (x = pred, fill= sex)) + geom_histogram () +
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
Zur Berechnung des RMSE kann auch die Funktion rmse() aus dem Paket performance verwendet werden. Hier wird manuell die Formel angewandt.
sqrt (mean ((d_train1$ pred - d_train1$ body_mass_g)^ 2 ))
Testen der Vorhersagegüte im eigenen Test-Sample:
<- d_train_test %>% mutate (pred = predict (lm1, newdata = d_train_test))sqrt (mean ((d_train_test$ pred - d_train_test$ body_mass_g)^ 2 ))
Für die eigene Abgabe trainieren wir unser bestes Modell noch einmal auf dem ganzen Train-Datensatz:
<- lm (body_mass_g ~ I (bill_depth_mm^ 2 ) + species + flipper_length_mm+ bill_length_mm + sex, data = d_train)summary (lm1)
Call:
lm(formula = body_mass_g ~ I(bill_depth_mm^2) + species + flipper_length_mm +
bill_length_mm + sex, data = d_train)
Residuals:
Min 1Q Median 3Q Max
-811.87 -155.90 -24.31 162.38 912.30
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -938.6799 604.1940 -1.554 0.121677
I(bill_depth_mm^2) 2.0075 0.6394 3.140 0.001917 **
speciesChinstrap -322.7564 92.4227 -3.492 0.000576 ***
speciesGentoo 968.6972 141.3086 6.855 6.71e-11 ***
flipper_length_mm 14.6584 3.3034 4.437 1.42e-05 ***
bill_length_mm 25.6652 8.4080 3.052 0.002541 **
sexmale 391.6317 56.1159 6.979 3.26e-11 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 280.4 on 226 degrees of freedom
Multiple R-squared: 0.883, Adjusted R-squared: 0.8799
F-statistic: 284.2 on 6 and 226 DF, p-value: < 2.2e-16
Berechnung der Note (für euch in der Klausur nicht möglich):
<- d_test %>% mutate (preds = predict (lm1, newdata = d_test))sqrt (mean ((d_test$ preds - d_test$ body_mass_g)^ 2 ))
Erstellung der Abgabe-Csv:
<- d_test %>% select (id, preds)write_csv (abgabe, "abgabe_notebook.csv" )