library(tidyverse)
library(visdat)16 Fallstudie Penguins Lösungen
Diese Fallstudie soll dazu dienen, den bisher gelernten Stoff sinnvoll auf einen Datensatz anzuwenden. Beschreibt in eurer Lösung am besten die einzelnen Schritte, die ihr ausführt, mit #Erklärungstext über dem Code. So ist euer Code nachvollziehbarer und in der Prüfung solltet ihr das genauso machen.

16.1 Pakete laden
Lade die erforderlichen Pakte (tidyverse, visdat)
16.2 Datenimport
Importiere den penguins Datensatz:
penguins <- read.csv("https://vincentarelbundock.github.io/Rdatasets/csv/palmerpenguins/penguins.csv")16.3 Datenjudo
Lass dir die obersten Zeilen ausgeben
penguins %>%
head()rownames <int> | species <chr> | island <chr> | bill_length_mm <dbl> | bill_depth_mm <dbl> | ||
|---|---|---|---|---|---|---|
| 1 | 1 | Adelie | Torgersen | 39.1 | 18.7 | |
| 2 | 2 | Adelie | Torgersen | 39.5 | 17.4 | |
| 3 | 3 | Adelie | Torgersen | 40.3 | 18.0 | |
| 4 | 4 | Adelie | Torgersen | NA | NA | |
| 5 | 5 | Adelie | Torgersen | 36.7 | 19.3 | |
| 6 | 6 | Adelie | Torgersen | 39.3 | 20.6 |
Verschaffe dir einen Überblick über die Variablentypen
penguins %>%
sapply(class) rownames species island bill_length_mm
"integer" "character" "character" "numeric"
bill_depth_mm flipper_length_mm body_mass_g sex
"numeric" "integer" "integer" "character"
year
"integer" Prüfe auf fehlende Werte
penguins %>%
summarise((across(everything(),~sum(is.na(.x)))))rownames <int> | species <int> | island <int> | bill_length_mm <int> | bill_depth_mm <int> | |
|---|---|---|---|---|---|
| 0 | 0 | 0 | 2 | 2 |
Prüfe die Variablen auf Ausreißer/Extremwerte
# Am Beipiel von flipper_length_mm:
ggplot(penguins, aes(flipper_length_mm))+
geom_boxplot()Warning: Removed 2 rows containing non-finite outside the scale range
(`stat_boxplot()`).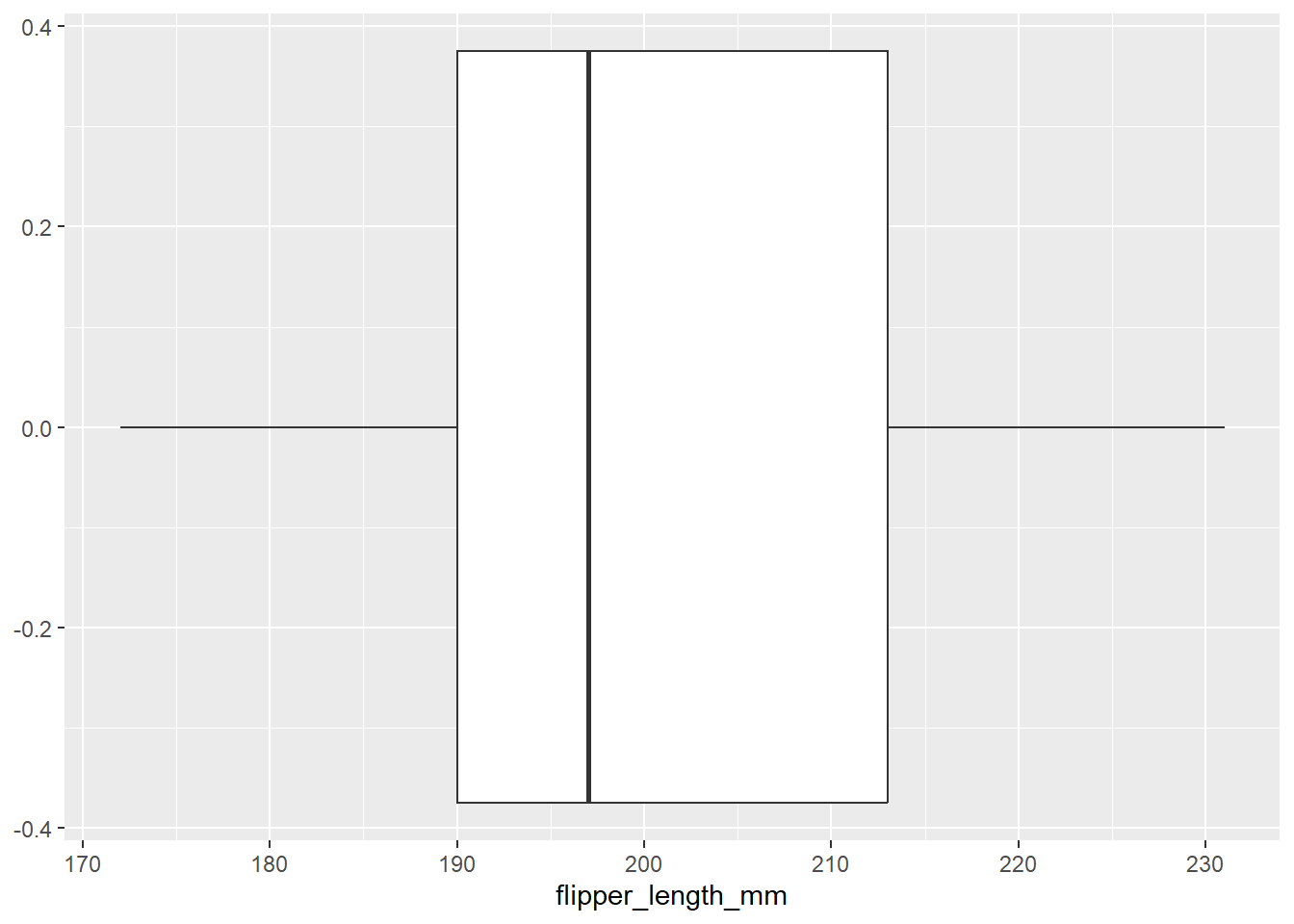
16.4 Datenvisualisierung
Plotte die Variablentypen
vis_dat(penguins)
Sieh dir die Verteilung von flipper_length_mm an
ggplot(penguins, aes(x = flipper_length_mm)) +
geom_histogram()`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.Warning: Removed 2 rows containing non-finite outside the scale range
(`stat_bin()`).
Sieh dir die Verteilung von flipper_length_mm in Abhängigkeit von der Spezies an
ggplot(penguins, aes(x = flipper_length_mm)) +
geom_histogram(aes(fill = species), alpha = 0.7)`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.Warning: Removed 2 rows containing non-finite outside the scale range
(`stat_bin()`).
Sieh dir die Verteilung der verschiedenen Spezies auf die unterschiedlichen Inseln an
ggplot(penguins, aes(x = island, fill = species)) +
geom_bar(alpha = 0.8) +
facet_wrap(~species, ncol = 1) +
coord_flip()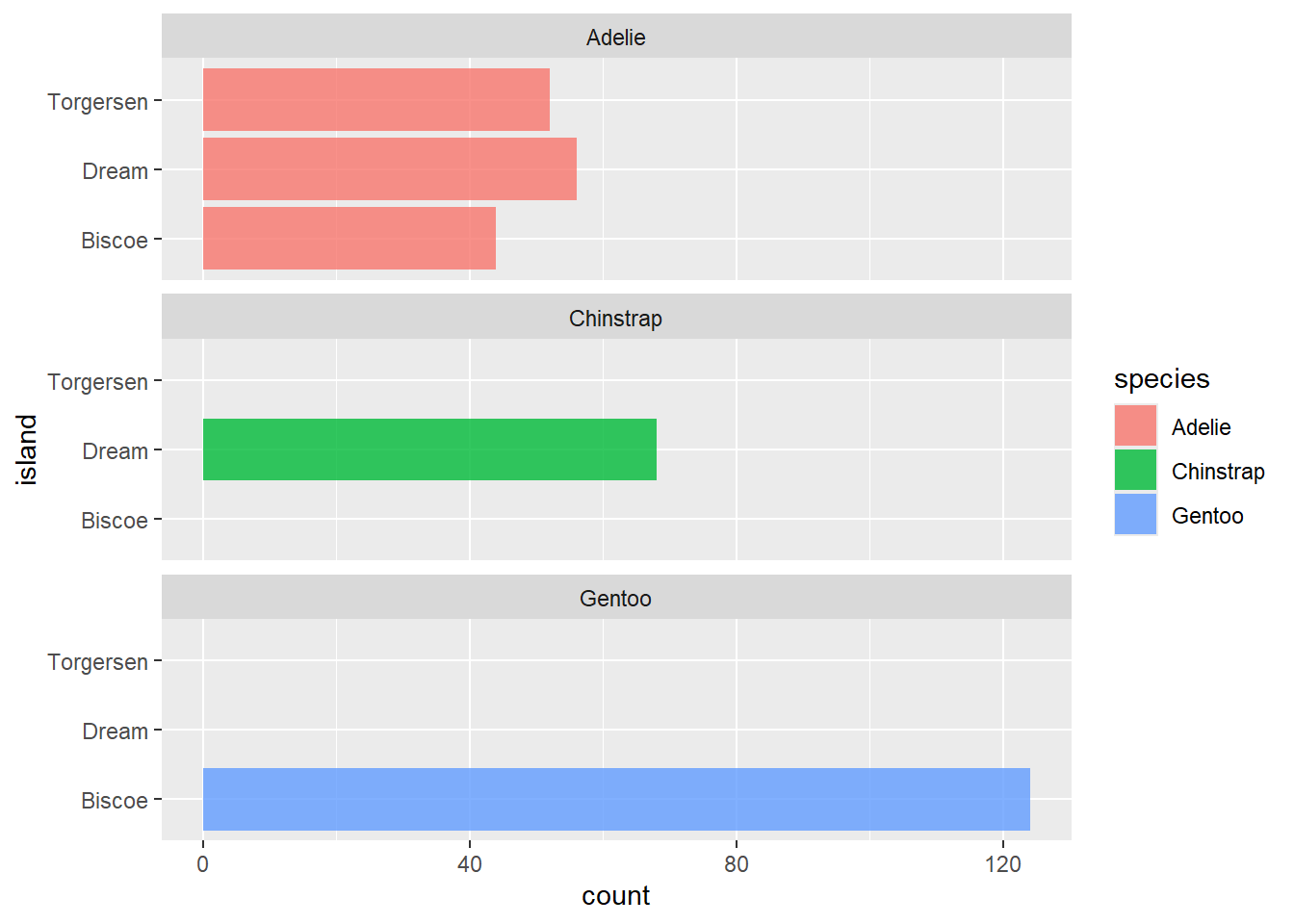
Sieh dir die Verteilung der Geschlechter unter den verschiedenen Spezies an
ggplot(penguins, aes(x = sex, fill = species)) +
geom_bar(alpha = 0.8) +
facet_wrap(~species, ncol = 1) +
coord_flip()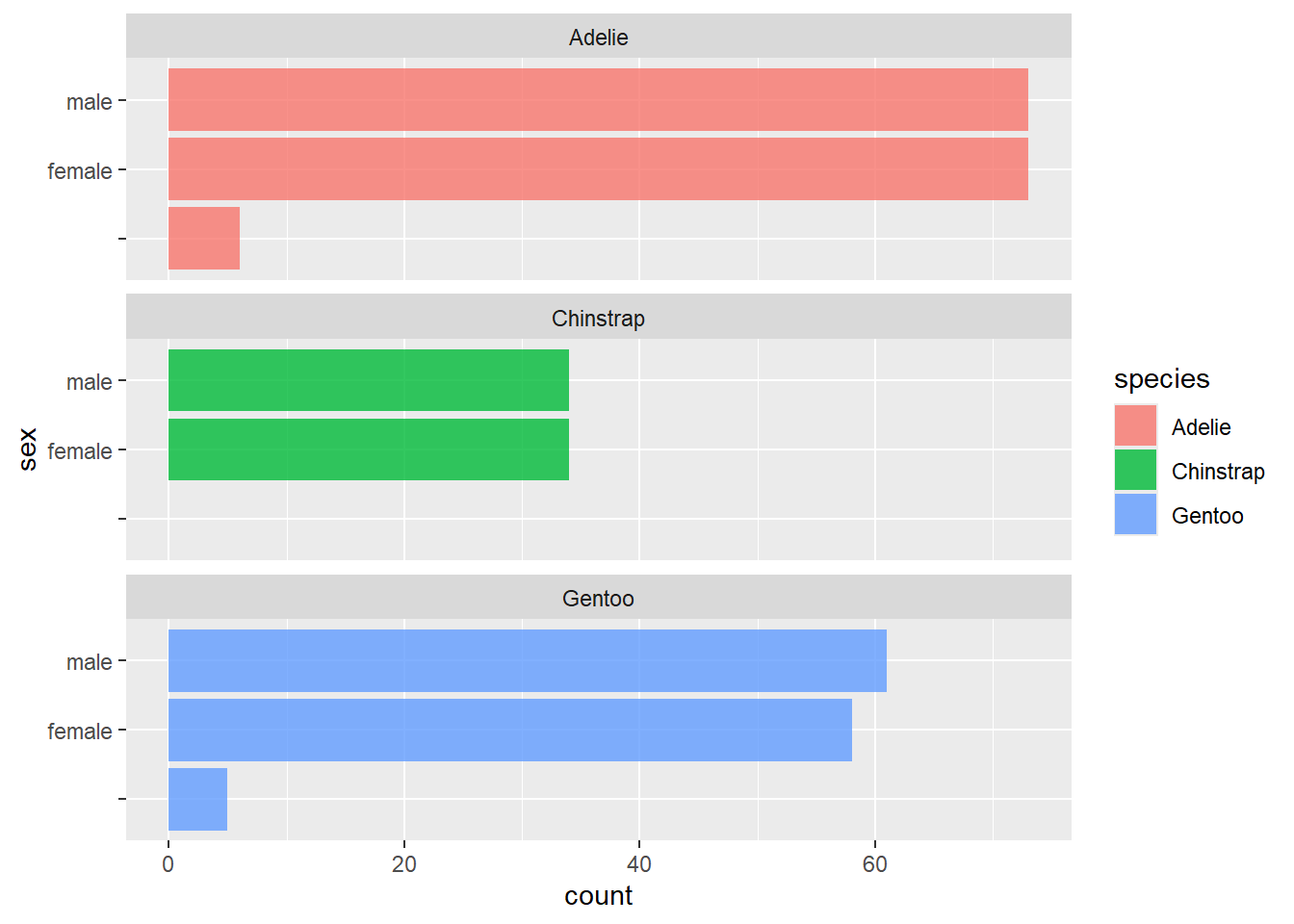
Sieh dir das Verhältnis von body_mass_g zu flipper_length_mm in Abhängigkeit der Spezies an und hebe, falls vorhanden, einen linearen Trend hervor
ggplot(data = penguins, aes(x = flipper_length_mm, y = body_mass_g)) +
geom_point(aes(color = species,
shape = species))+
geom_smooth(method = lm)`geom_smooth()` using formula = 'y ~ x'Warning: Removed 2 rows containing non-finite outside the scale range
(`stat_smooth()`).Warning: Removed 2 rows containing missing values or values outside the scale range
(`geom_point()`).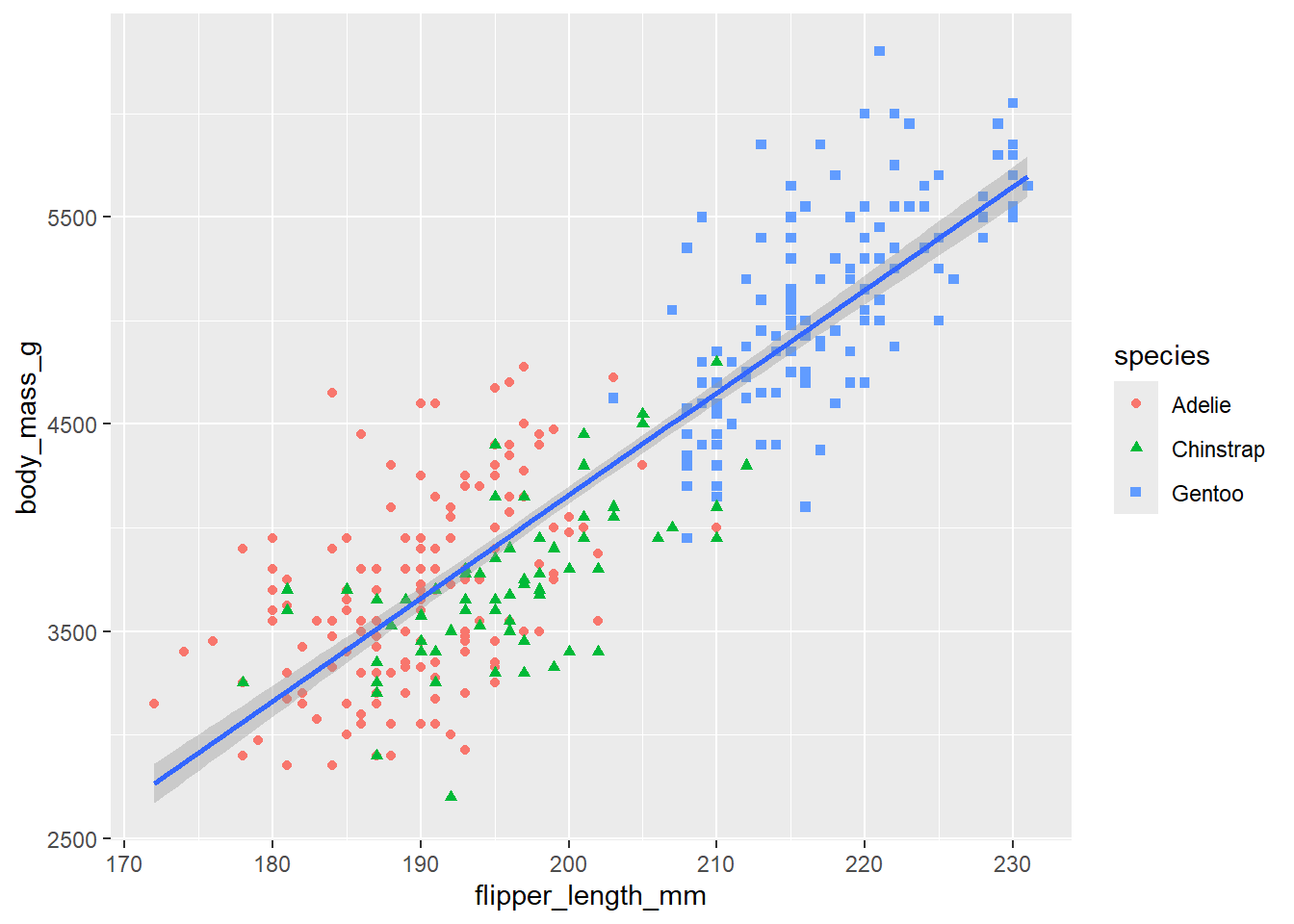
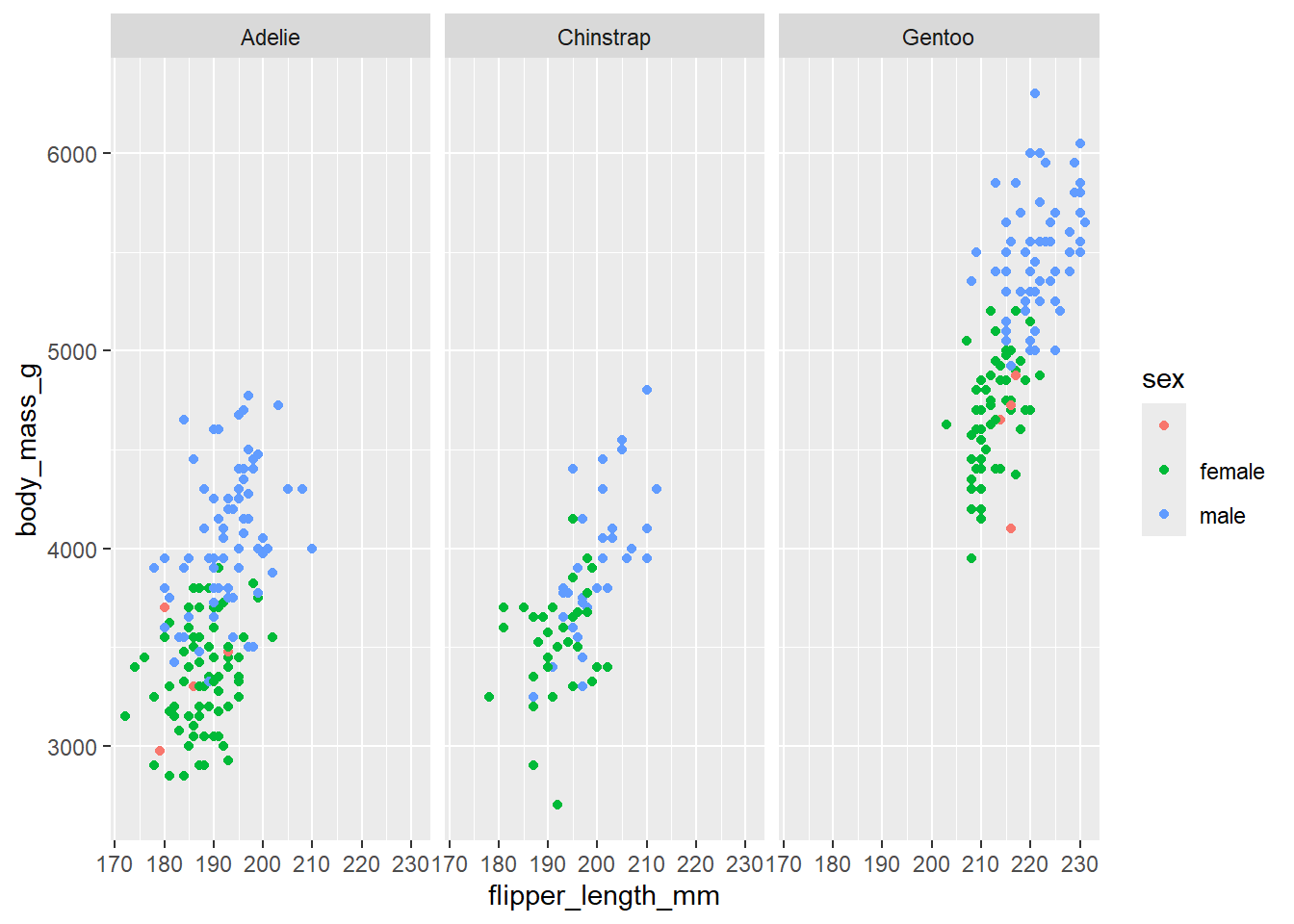
Sieh dir das Verhältnis von body_mass_g zu flipper_length_mm in Abhängigkeit des Geschlechts für jede Spezies an
ggplot(penguins, aes(x = flipper_length_mm,
y = body_mass_g)) +
geom_point(aes(color = sex)) +
facet_wrap(~species)Warning: Removed 2 rows containing missing values or values outside the scale range
(`geom_point()`).